由于mRNA-seq测出来的是mRNA在细胞中的数量,而最终在细胞中发挥功能的却是蛋白质,因此mRNA的数量并不能很好的反应细胞的生长状态。目前科学家发现mRNA水平与细胞表型的相关性只有约0.13~0.16左右。为此,科学家开发了Ribo-seq技术,用于表征不同mRNA的不同翻译状态。
高分辨率Ribo-seq(Ribosome Profiling)技术通过捕获并测序核糖体保护片段(Ribosome-Protected Fragments, RPFs),实现了在单核苷酸分辨率下对mRNA翻译过程的全景式观察。自其问世以来,Ribo-seq已成为解析转录后调控、识别翻译起始位点、监测翻译速率变化乃至揭示新型编码序列(如uORFs、smORFs)的核心技术手段。尤其是在疾病状态下,如肿瘤或病毒感染过程中,Ribo-seq能够提供传统转录组学无法获取的翻译层级信息,从而揭示调控失衡的分子机制。
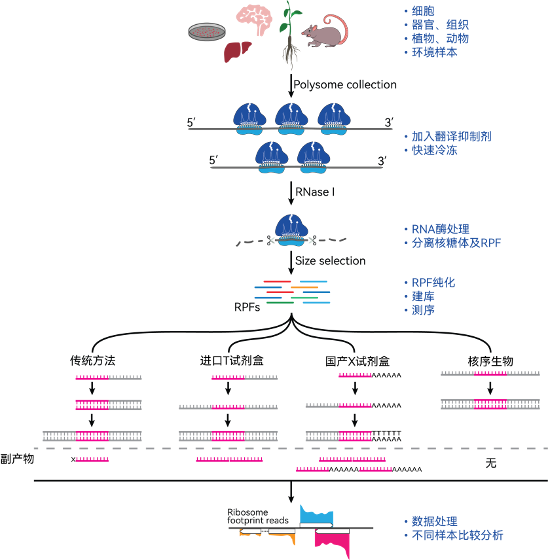
图1、Ribo-seq的实验原理示意图
Ribo-seq的实验原理如图 1所示,当核糖体在mRNA上翻译时,小分子药物的加入可使翻译暂停,使得核糖体停留在mRNA上而不掉下来。真核细胞常使用Cycloheximide(CHX,放线菌酮/环己酰亚胺),原核生物常使用氯霉素。当核糖体以及所翻译的mRNA被提取出来之后,通过RNase的酶解,可以去除掉mRNA。但是其中被核糖体保护的片段(Ribosome protected fragment,RPF)无法被RNase酶解。不同物种由于核糖体大小不一样,RPF长度也不一样,通常在27~31nt。
由于RPF是受核糖体保护留下的片段,因此RPF能够代表核糖体在mRNA上的位置,RPF的数量也能代表mRNA上核糖体的数量。针对RPF进行高通量测序,就能够表征mRNA上核糖体的位置和数量,也就是mRNA的翻译状态。
Ribo-seq可解决的问题
1. 表征基因对蛋白水平的调控。
由于RPF的数量就是翻译中的核糖体的数量,而每个核糖体能够翻译出一条多肽链(蛋白),因此RPF的数量就能够代表翻译出的蛋白质的数量。当将mapping到某一转录本(mRNA)上的所有RPF进行加和,得到这一mRNA的RPF数量。比较control细胞与gene KO细胞中mRNA上RPF的数量,可以筛选出RPF存在差异的基因,也就能代表某一gene KO能够调控其他基因蛋白水平的上调或下调。
2. 表征基因对翻译效率的调控。
Ribo-seq与mRNA-seq存在一个不同点:由于细胞中DNA的拷贝数是恒定的,因此mRNA-seq所获得的reads数不仅能够代表细胞中mRNA的数量,还能够代表基因的转录效率。但是由于每个细胞中mRNA的拷贝数不固定,因此mRibo-seq所获得的RPF只能代表蛋白的数量。
为了表征翻译效率的调控,Ribo-seq通常与RNA-seq同时使用。将一条mRNA上通过Ribo-seq获得的RPF数量除以通过RNA-seq获得的reads数,即可得到TE值(Translation efficiency,翻译效率)。由此可见,通过联合使用Ribo-seq与mRNA-seq,我们能够同时计算出基因的转录效率、翻译效率以及最终产生的蛋白质水平。
3. 不同细胞中翻译状态的改变。
上述我们通过TE可以表征翻译效率的变化,但翻译过程是复杂的,翻译效率的改变可以发生在翻译起始、翻译延伸以及翻译终止的任一过程,因此表征mRNA的真实翻译状态尤为重要。最典型的例子是癌细胞中“核糖体停滞”现象大大增加,但是这些基因在整体翻译效率上却没有显著(大于2倍)的差异。因此在翻译效率的基础上进一步研究不同的翻译状态能够更好的表征细胞的生长情况,研究新的细胞调控机制。
4. 发现新的开放阅读框(ORF)。
由于RPF代表核糖体在mRNA上的位置,因此对一些低丰度RPF进行标注,可以在ncRNA上发现一些新的ORF,或者在5‘UTR上发现新的uORF。近年来,一些通过Ribo-seq技术发现的ORF已经被质谱或其他方法证明。
Ribo-seq相关文献
1. Molecular Cell:科学家通过Ribo-seq发现核糖体通过广泛移码和框外mRNA降解调控转录组丰度。
2025年5月15日,瑞典卡罗林斯卡医学院VicentPelechano团队在Molecular Cell上发表了一篇题为“Ribosomes modulate transcriptome abundance via generalized frameshift and out-of-frame mRNA decay”的论文,发现在营养缺乏时,酿酒酵母大量转录本发生核糖体−1移码并被协同翻译机制加速降解。该过程由低密码子优化性驱动,且在多种生物中进化保守,揭示了一种耦合蛋白需求与mRNA丰度、限制细胞生长的反馈机制,拓展了mRNA质量控制的功能。
2. Science:胰腺癌特异性隐性抗原是T细胞识别的靶标
2025年5月8日,麻省理工William A.Freed-Pastor团队在Science上发表了一篇题为“Pancreatic cancer–restricted cryptic antigens are targets for T cell recognition”的论文,通过患者来源类器官富集胰腺癌细胞,鉴定出大量HLA结合的隐匿肽。研究开发了基于翻译的筛选策略,并在健康组织中排除非特异性翻译产物,筛出具肿瘤特异性的ncHLAp,并诱导识别它们的T细胞及TCR,验证其在临床前模型中可杀伤癌细胞。
3. Cell:m6A通过改变核糖体动态以启动mRNA降解
2025年5月5日,美国康奈尔大学Samie R.Jaffrey团队在Cell上发表了一篇题为“m6A alters ribosome dynamics to initiate mRNA degradation”的论文,发现m6A通过诱导核糖体停滞和碰撞,改变翻译动力学并促进mRNA降解。揭示核糖体是感应m6A并启动其介导降解的关键环节,也解释了m6A-mRNA在基础状态下表达水平较低,而在细胞应激期间表达升高的原因。
4. Cancer Cell:核糖体“叛变”——蛋白质工厂助推癌症进展
2025年4月24日,美国纽约洛克菲勒大学Sohail F. Tavazoie和QiushuangWu在Cancer Cell上发表了一篇题为“Ribosomal rebellion: When protein factories drive cancer progression”的评论文章。阐述Kuzuoglu-Ozturk等人发现eIF4A介导的促癌转录本翻译起始增强驱动了癌症进展。而Weller等人则揭示了异常的tRNA修饰如何破坏翻译准确性,从而产生新抗原。
5. AJHG :翻译调控与唇腭裂——tRNA剪接缺陷如何影响神经脊细胞发育。
2025年4月17日,美国麻省理工生物系EliezerCalo团队在The American Journal of Human Genetics上发表了一篇题为“A non-syndromic orofacial cleft risk locus links tRNA splicing defects to neural crest cell pathologies”的论文,发现DDX1是tRNA剪接复合物的关键组分,其在cNCCs中的缺失会导致tRNA剪接障碍,进而引发核糖体停滞、蛋白合成受阻和细胞迁移受损,最终干扰颅面发育。研究揭示了tRNA剪接缺陷导致口面裂的机制,并确认MYCN、DDX1及tRNA加工异常为致病风险因素。